在做机器学习模型的时候,一定要先做特征选择,其中用相关性做特征选择是一个非常常见的做法。但在这个看似简单的做法里其实有很多问题。这篇文章就和大家一起讨论一个相关性特征筛选的坑。
相关性特征选择方法一般是这样的:如果特征A和特征B的相关性大于某个阈值,则把区分度较差的那个特征剔除。这个方法看上去人畜无害,但用在时间切片型特征上会有个很有意思的现象。
我们以个人征信的数据为例来解释。
个人征信的数据有很多是时间序列,比如一个用户在30天内的登录次数,60天内的登录次数,90天内的登录次数等。这种按时间切片来构造的特征天生的相关性就很高,因为计算逻辑都是一样的,只是时间跨度有区别。但短时间和长时间的数据又具有各自的特点:
- 短时间数据:反应用户短期的行为变化
- 长时间数据:反应用户长期行为的稳定性
假设我们有如下特征,他们的区分度和相关性分别如下:
区分度
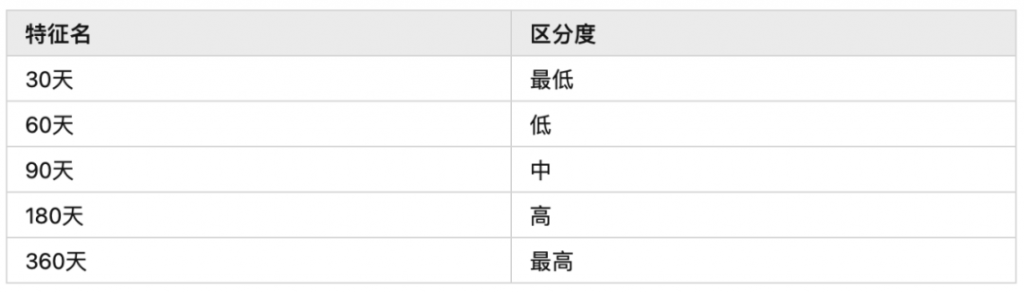
(一般时间切片的特征时间周期长区分度会高)
相关性
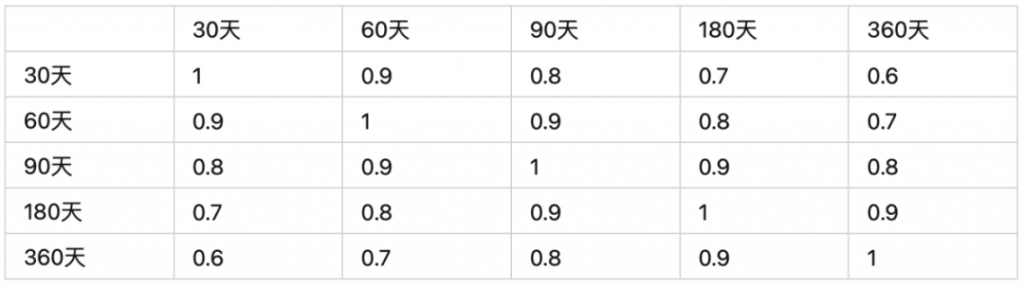
我们按照上面描述的相关性筛选方法来做一遍特征选择,这里我们假设相关性阈值设为0.9。
第一步:我们首先拿出30天和60天的特征,发现相关性达到0.9,则在30天和60天两个特征内剔除区分度低的,这里30天特征的区分度低,则剔除30天的特征。
第二步:继续拿60天和90天的特征比,相关性满足0.9的阈值,然后再60天和90天特征里剔除区分度低的特征,则60天的特征被剔除。
第三步:接着拿90天和180天的特征比,大家应该可以感觉到了,90天的特征也会被剔除。
第四步:最后拿180天和360天的特征比,也会剔除180天的特征,最后仅仅剩下360天的特征。
这个筛选很明显是有问题的。
30天和360天的特征的相关性其实并不高,这两个特征是可以并存的。但因为我们是按照区分度从低到高两两比较特征,在时间切片的特征构造逻辑下,两个相近的时间切片上的特征相关性一定是高的。这就会发生上述的链式剔除的效应,把本来可以共存的特征都剔除了。
那如何来解决这个相关系筛选的问题呢?
我们其实只需要做一个简单的调整,从剔除特征改为选入特征:把特征按区分度从高到低排列,先选入区分度最高的特征,然后按区分度从高到低的选入特征,选入的标准是当这个特征和所有已经选入的特征相关性小于某阈值时才可选入。
我们拿上面的例子演示一遍。
第一步:首先把特征按区分度从高到低排列,选入区分度最高的360天的特征。
第二步:按区分度从高到低查看特征,那现在我们会查看180天的特征,因为在剩余特征中,他的区分度最高,然后看180天的特征和360天的特征相关性是否小于阈值,这里假设阈值是0.9,则不满足,继续看后面的特征。
第三步:继续查看90天的特征,他和360天的特征相关性小于0.9,则选入这个特征,这样我们的特征集就有360天和90天的特征了。
第四步:继续查看60天的特征,60天的特征和360天的特征相关性小于0.9,满足阈值要求,但是60天和90天的相关性不满足阈值要求,pass。
第五步:继续查看30天的特征,30天和360天的特征相关性满足阈值要求,30天和90天也满足阈值要求,则选入特征集,算法停止。
这样按相关性选入的特征就有30天,90天和360天三个特征,包含了短时间和长时间的特征。反观之前的算法选出来的特征,只有360天一个长时间的特征。虽然这两个相关性做特征筛选的逻辑很相似,但在实际的时间切片数据上,实际性能还是有很大区别的。
诸如此类模型面试与工作的各种坑点,以及应对经验方法,我会在风控模型训练营里为大家详细讲解,例如第一周课上会讲到的7种特征选择方法全总结,具体可以咨询美女科科老师

最后,附上相关性特征选择的代码供大家参考:(更多模型代码模版,我也会在模型训练营与大家实操共享)
import numpy as np
import pandas as pd
class Metrics:
@classmethod
def get_iv(cls, df_label, df_feature):
var = df_feature.name
df_data = pd.DataFrame({'val': df_feature, 'label': df_label})
# statistics of total count, total ratio, bad count, bad rate
df_stat = df_data.groupby('val').agg(total=('label', 'count'),
bad=('label', 'sum'),
bad_rate=('label', 'mean'))
df_stat['var'] = var
df_stat['good'] = df_stat['total'] - df_stat['bad']
df_stat['total_ratio'] = df_stat['total'] / df_stat['total'].sum()
df_stat['good_density'] = df_stat['good'] / df_stat['good'].sum()
df_stat['bad_density'] = df_stat['bad'] / df_stat['bad'].sum()
eps = np.finfo(np.float32).eps
df_stat.loc[:, 'iv'] = (df_stat['bad_density'] - df_stat['good_density']) * \
np.log((df_stat['bad_density'] + eps) / (df_stat['good_density'] + eps))
cols = ['var', 'total', 'total_ratio', 'bad', 'bad_rate', 'iv', 'val']
df_stat = df_stat.reset_index()[cols].set_index('var')
return df_stat['iv'].sum()
class CorrSelector:
def __init__(self):
self.detail = dict()
self.selected_features = list()
self.removed_features = list()
def fit(self, df_xtrain, df_ytrain, **kwargs):
corr_threshold = kwargs.get('corr_threshold', 0.95)
method = kwargs.get('method', 'iv_descending')
n_jobs = kwargs.get('n_jobs', -1)
feature_list = sorted(kwargs.get('feature_list', df_xtrain.columns.tolist()))
lst_iv = [Metrics.get_iv(df_ytrain, df_xtrain[c]) for c in feature_list]
df_iv = pd.DataFrame({'var': feature_list, 'iv': lst_iv})
df_iv = df_iv.sort_values(by='iv', ascending=False).set_index('var')
feature_list = df_iv.index.tolist()
df_corr = df_xtrain[feature_list].corr()
df_corr = abs(df_corr - pd.DataFrame(np.identity(len(df_corr)),
index=df_corr.index,
columns=df_corr.columns)) # remove self
df_res = pd.concat([df_iv, df_corr], axis=1)
self.detail['before'] = df_res
for var in feature_list:
if var not in df_res.index:
continue
else:
lst_remove = df_res[df_res[var] >= corr_threshold].index.tolist()
df_res = df_res.drop(lst_remove, axis=0)
df_res = df_res.drop(lst_remove, axis=1)
self.detail['after'] = df_res
self.selected_features = df_res.index.tolist()
self.removed_features = sorted(set(feature_list) - set(self.selected_features))
return self
def transform(self, df_xtest, **kwargs):
feature_list = kwargs.get('feature_list', df_xtest.columns.tolist())
feature_list = sorted(set(feature_list) & set(self.selected_features))
return df_xtest[feature_list]
